近日,中国农业科学院北京畜牧兽医研究所微生物与酶工程科技创新团队基于蛋白质大语言模型,开发出新型多功能抗菌肽从头设计方法DLFea4AMPGen。该方法可高效筛选并生成具有多类生物活性的功能肽,在智能化抗菌肽设计与开发方面展现出巨大潜力。相关研究成果发表在《自然通讯(Nature Communications)》上。
抗菌肽是广泛存在于动植物和微生物体内的天然防御分子,能够通过物理破坏细菌膜来规避现有耐药机制,已成为应对抗生素耐药性威胁的重要候选药物。然而,当前抗菌肽的从头设计策略仍存在设计成功率低、依赖大规模虚拟肽库构建等问题,严重制约其开发效率与应用可行性。
研究人员基于其团队构建的蛋白质大语言模型MP-TRANS,开发了系列活性肽的预测工具,实现通用蛋白质表征向抗菌肽设计任务的高效迁移。通过进一步引入SHAP可解释性分析,定量评估每一残基对抗菌、抗真菌及抗氧化活性的贡献,识别出高贡献特征片段,并通过重组构建出候选肽序列子空间。实验验证显示,筛选的16条肽中,有12条肽具备两种及以上生物活性,其中候选肽D1在体内外均表现出对多重耐药菌的显著抑制效果。该研究实现了“少样本、高精度”的抗菌肽理性设计,彰显了蛋白大语言模型与国产高性能计算在推动活性肽研究创新中的重要作用。
中国农业科学院北京畜牧兽医研究所博士研究生高翰、生物技术研究所关菲菲博士为论文共同第一作者,牧医所罗会颖研究员、田健研究员、黄火清研究员为论文共同通讯作者。该研究得到国家重点研发项目、自然科学基金、中国农业科学院科技创新工程等项目的资助,同时还得到了其他合作单位的支持。
原文链接:https://www.nature.com/articles/s41467-025-64378-y
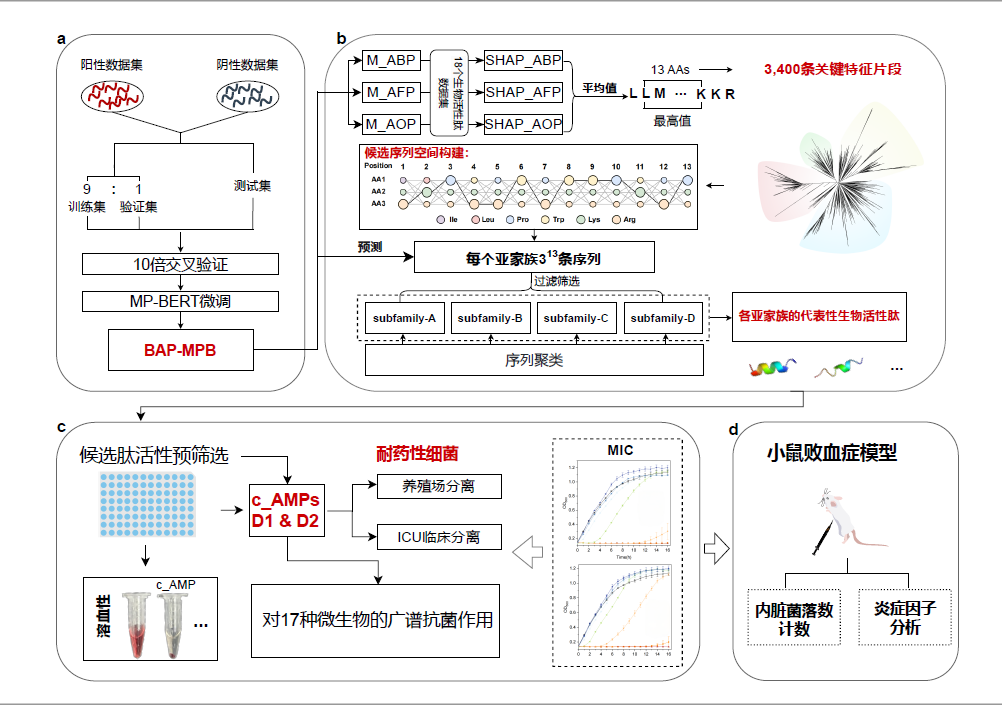
图 DLFea4AMPGen 的设计流程与实验验证工作框架










