近日,中国农业科学院北京畜牧兽医研究所奶产品质量与风险评估科技创新团队成功建立了一种分析瘤胃微生物脲酶的靶向宏蛋白质组方法,为进一步揭示瘤胃脲酶蛋白多样性和发展完善靶向宏蛋白质组学方法提供了指导。相关研究成果发表在《微生物前沿(Frontiers in Microbiology)》上。
宏蛋白质组学方法是研究微生物群落功能的重要技术方法,对于连接上游宏基因组和下游宏代谢组具有重要意义。靶向宏蛋白质组方法是指针对某一类功能微生物的某个或多个蛋白或酶,通过宏蛋白质组学方法体系,研究某一类功能微生物的多样性与功能调控的方法,具有功能靶向性强、表型关联性高和调控机制明显等优势。但是,目前靶向宏蛋白质组方法应用的较少,尤其是在瘤胃微生物功能研究方面鲜有报道。脲酶是瘤胃微生物生态系统中重要的酶,在尿素氮循环利用和微生物蛋白质合成中扮演重要角色,但是目前仍缺乏蛋白质水平的脲酶多样性研究。
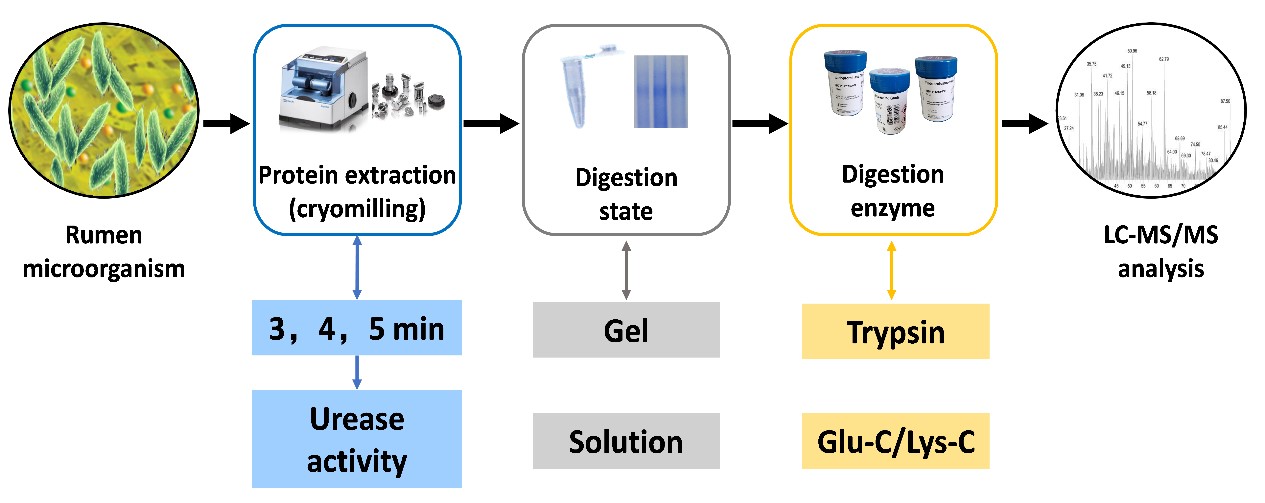
为了构建瘤胃微生物脲酶的靶向宏蛋白质组方法,研究人员通过检测脲酶活性,优化了奶牛瘤胃微生物蛋白质提取(冷冻珠磨时间)条件,分析了瘤胃微生物高效酶解方法(胶条、溶液),优化了适合脲酶蛋白的消化酶种类(胰蛋白酶、蛋白内切酶谷氨酸-C、蛋白内切酶赖氨酸-C)。结果表明,液氮冷冻珠磨4分钟时可提取到最高脲酶活性的蛋白质,胶内结合胰蛋白酶酶解方式能鉴定到最多的脲酶肽段。利用已知脲酶蛋白数据库进行鉴定分析,发现瘤胃中存在大量新的未被认识的脲酶蛋白,并且脲酶蛋白多样性较高。在鉴定的脲酶蛋白中,发现他们主要来自于原绿球菌和螺杆菌等。
该研究得到国家重点研发计划、中国农业科学院重大任务和现代农业产业技术体系等项目资助。张晓音为文章第一作者,赵圣国和王加启为文章通讯作者。










