近日,牧医所利用高密度SNP芯片数据对我国不同尾型的地方绵羊品种进行了有效群体大小估计、全基因组长纯合片段(ROH)扫描及高频ROH基因组区段鉴定,检测出不同群体特有和共有的ROH基因组区段,为保护和开发利用我国地方绵羊品种、深度挖掘功能基因提供了重要参考。相关成果发表在《畜牧与生物技术杂志(Journal of Animal Science and Biotechnology)》(IF=5.032)上。
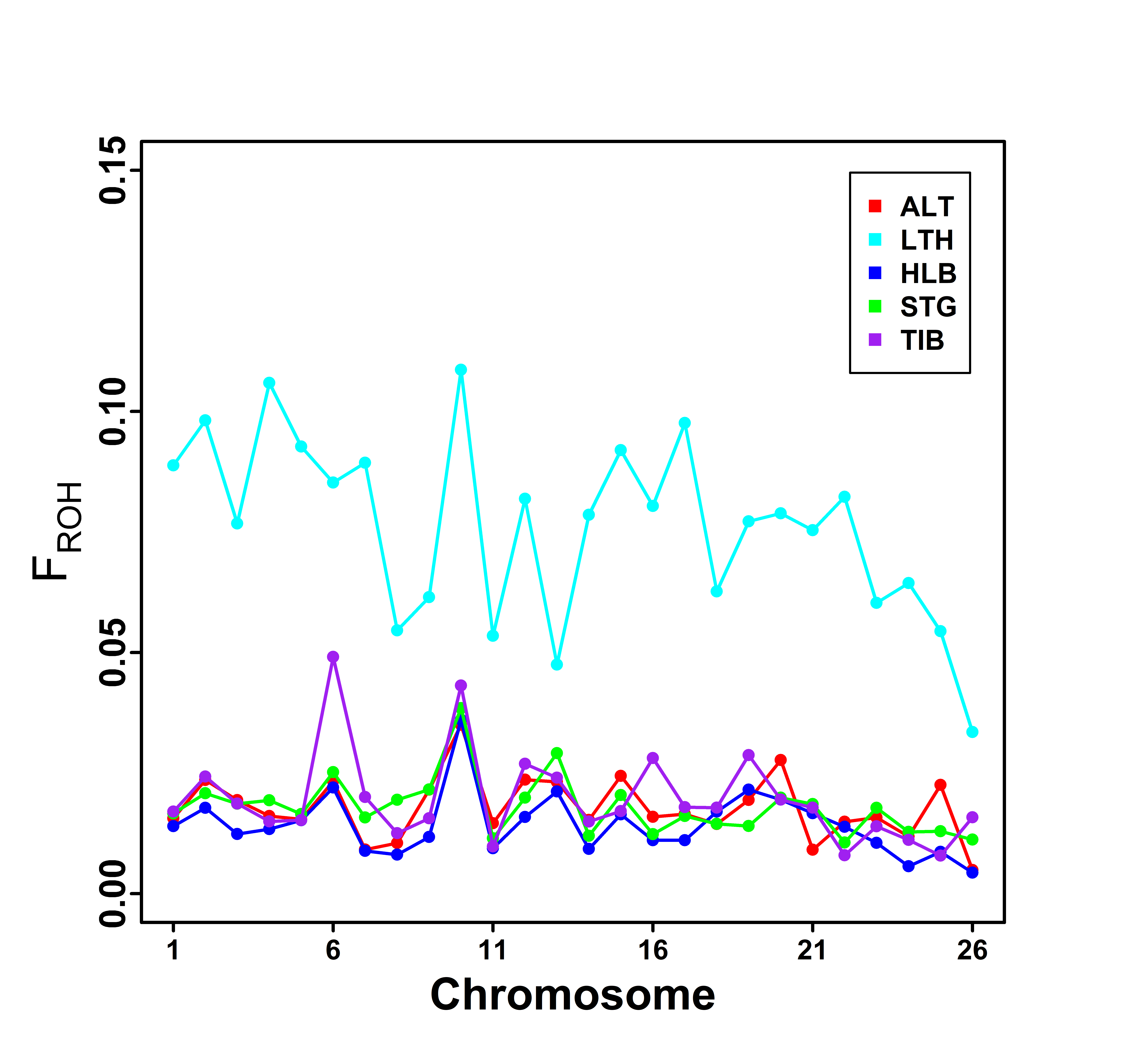
据王立贤研究员介绍,良好的近交控制是畜禽遗传改良和畜牧业可持续发展的重要保障。传统的利用系谱信息评估近交(FPED)在实际应用中存在系谱信息难收集、错误率高、历史近交被忽略等问题。随着全基因组测序和基因芯片等成本的降低,利用分子信息评估近交成为研究的热点。研究人员利用高密度SNP芯片数据(600k)对大尾寒羊,阿拉泰羊,呼伦贝尔羊、草原短尾羊和西藏羊等不同尾型的五个绵羊品种进行了连锁不平衡检测、有效群体大小估计、观测杂合度和期待杂合度近交系数计算(FHOM),基于ROH进行了全基因组近交系数计算(FROH)。结果显示,多种遗传多样性评估结果与FROH评估结果几乎一致。特别是基于ROH检测到大尾寒羊群体最高的平均近交水平达到0.0808,并且具有最高比例的ROH片段(0.037),表明该群体近世代有效群体小、近交程度高,与大尾寒羊的群体现状相吻合。此外,研究人员基于不同群体内高频ROH区域,筛选出了与群体特征一致的重要经济性状基因,例如与绵羊尾部脂肪沉积相关的 PDGFD 、 HOXA10 基因,与生长发育相关的 TNNI1 、 CSRP1 和 EEF1A2 基因。

该研究得到国家自然科学基金和中国农业科学院科技创新工程等项目的支持。硕士研究生刘家鑫和博士研究生史良玉为文章共同第一作者,王立贤研究员和赵福平研究员为共同通讯作者。
原文链接:https://jasbsci.biomedcentral.com/articles/10.1186/s40104-021-00608-9










